大家好,我是邓飞,之前推荐过这本《Genome-Wide Association Studies》的书籍,2022年出版的,内容比较前沿。
这几天看过一遍了,觉得干货很多,有很多知识点的连通,弄懂了之前是是而非的概念,收获很大。我觉得阅读一本书,如果把刚读懂的输出写篇文章,或者把没有搞懂的内容写出来交流一下,是掌握知识的快捷方法。所以,我计划,把每一章节搞成一篇文章。主要内容,不是单纯的翻译,而是拆解之后的再学习。
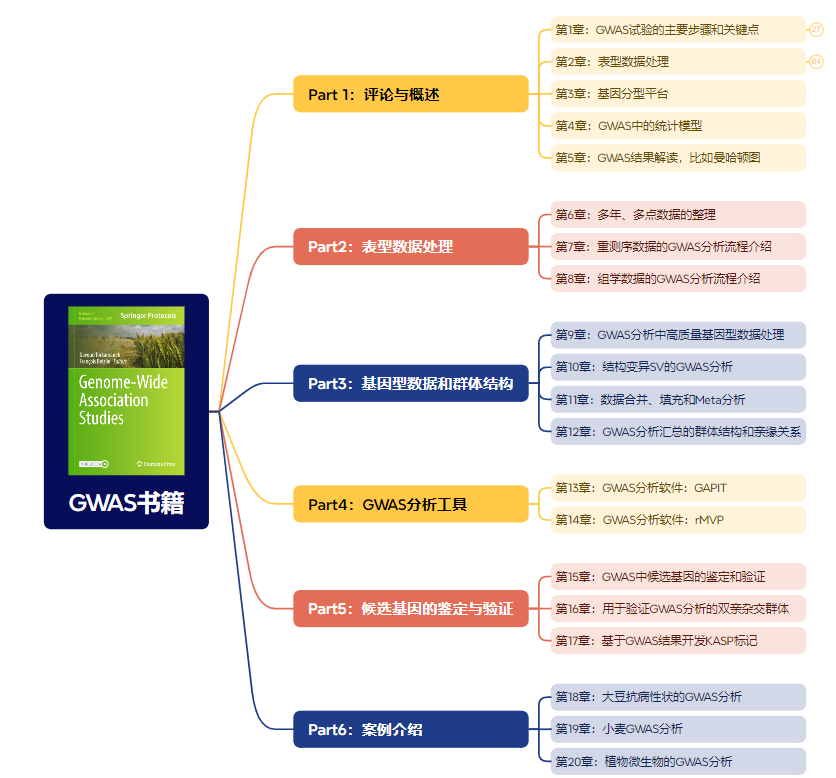
这本书的目录:
今天介绍第一章:《GWAS分析的主要步骤和关键要点》。
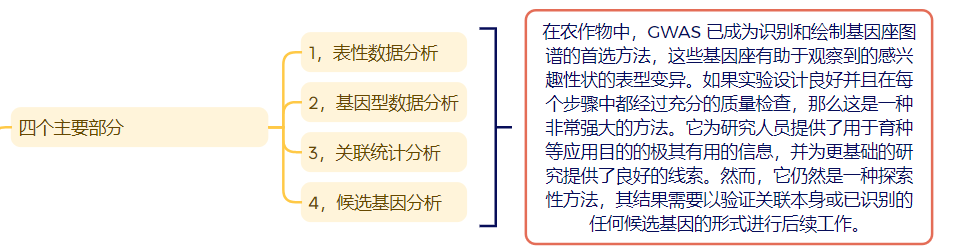
GWAS分析大体有四个部分:
-
1,表型数据分析
-
2,基因型数据分析
-
3,关联分析
-
4,候选基因分析
GWAS VS QTL:
-
QTL都是人工群体,有高度的LD衰减,所以需要比较少的标记就可以。但是QTL缩小区间,需要大量的群体才能找到交换单株,工作量巨大。
-
GWAS群体LD衰减距离很快,所以需要高密度的标记。GWAS成为育种群体挖掘基因的更有利工具。进而应用于MAS。
-
在多基因效益以及基因间互作的复杂遗传结构,GWAS通常可以提供相对于双亲图谱更高的遗传分辨率
SV分析GWAS的思路:
-
SV也可以变为主等位纯合、杂合、次等位纯合的格式,编码为0-1-2的格式,用GAPIT分析完全没问题。
-
研究表明,SV的GWAS结果和SNP的GWAS结果,重复度在93%以上。同时,7%是SV单独解释的,SV解释抗病性中更大比例的变异。
GWAS中考虑单倍型
-
填充,低密度填充到高密度,需要用到单倍型
-
定位基因时,单倍型数据可以降低假阳性
GWAS分析汇总不同模型共有位点:
-
有共有位点,说明这个位点可信度比较高
-
没有共有位点,并不意味着假阳性或者错误的结果,还需要进一步验证。
做GWAS的自我灵魂拷问:

下一篇,介绍《表型数据处理》,欢迎继续关注。
